4.1 Wie finden wir neue Enzyme für industrielle Anwendungen?
Um Enzyme in großer Menge für die industrielle Anwendung produzieren zu können, werden optimierte Produktionsstämme verwendet. Diese stellen das gewünschte Enzym her. Sie benötigen dafür den Bauplan des Proteins – das Gen. Wie gelangen wir also an die Baupläne von neuen unbekannten Enzymen?
DNA-Isolierung
Der Bauplan von Proteinen, also die Gene, sind in der DNA kodiert. Wir benötigen also die DNA der Organismen, die Gene für neue unbekannte Enzyme enthalten. Um die DNA aus den Zellen zu isolieren, werden die Zellen aufgebrochen und die gesamte darin enthaltene DNA von den restlichen Zellbestandteilen (Proteine, Membranen,…) abgetrennt.
Die DNA kann dann zur Größenanalyse mittels Agarose-Gelelektrophorese aufgetrennt werden. Wie das funktioniert seht ihr hier.
Wie kann man mit Haushaltschemikalien eine DNA-Isolierung aus Tomaten vornehmen?

Schaut es euch in diesem Film. an!
Video: Biokatze (2012). Isolierung der DNA aus Tomaten.
Link: https://youtu.be/_5rm8r2RJJw
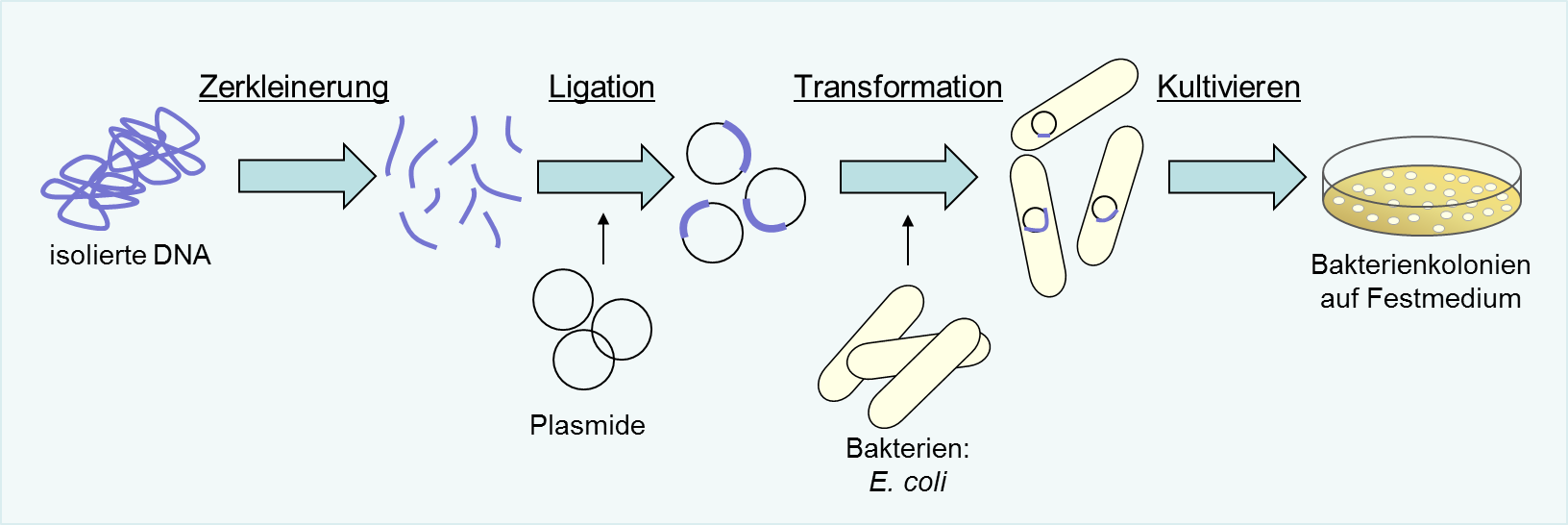
Erstellung einer Genbank
Abb.: Erstellung einer Genbank
Um in der großen Menge an DNA mit sehr vielen unterschiedlichen Informationen das gewünschte Gen zu finden, wird eine Genbank erstellt. Hierfür wird die DNA zunächst in Fragmente zerkleinert. Dies kann durch mechanische Kräfte oder durch sogenannte Endonukleasen erfolgen. Endonukleasen sind Enzyme und schneiden die DNA bei bestimmten Erkennungssequenzen.
Die DNA-Fragmente werden anschließend in ringförmige DNA-Moleküle, sogenannte Plasmide, eingebracht. Diesen Vorgang nennen wir Ligation. Jedes der erzeugten Plasmide enthält im Optimalfall ein anderes Stück DNA.
Um die auf den DNA-Fragmenten enthaltenen Informationen in Proteine umzuwandeln, werden die fertigen Plasmide in Bakterien eingebracht. Diesen Schritt nennen wir Transformation. Hierfür verwenden wir meist das gut erforschte Bakterium E. coli. Das Einbringen der Plasmide in E. coli kann dabei über einen Hitze- oder Elektroschock (Elektroporation) erfolgen.
Die Bakterien werden anschließend auf festem Nährmedium verteilt und kultiviert. Dabei teilen sich die Bakterien, sodass kleine Zellhaufen entstehen, die wir Kolonien nennen. Die Zellen einer Kolonie enthalten dabei alle dasselbe Plasmid. Jede Kolonie enthält idealerweise ein anderes Plasmid mit einem anderen DNA-Fragment. Unsere Genbank ist fertig!
Das Nährmedium, auf dem die Zellen kultiviert werden, enthält ein Antibiotikum. Es können nur Zellen wachsen, die ein entsprechendes Resistenzgen enthalten. Dieses Resistenzgen ist in allen unserer Plasmide enthalten. Es überleben also nur die Zellen, die auch eines der Plasmide enthalten. Zellen behalten Plasmide und geben sie auch nur an ihre Tochterzellen weiter, wenn sie ihnen einen solchen entscheidenden Vorteil bieten. Plasmide sind für Zellen nämlich Schwerstarbeit: Bei jeder Zellteilung müssen auch die Plasmide vervielfältigt werden.
Screening: Die Suche nach dem „richtigen“ Gen
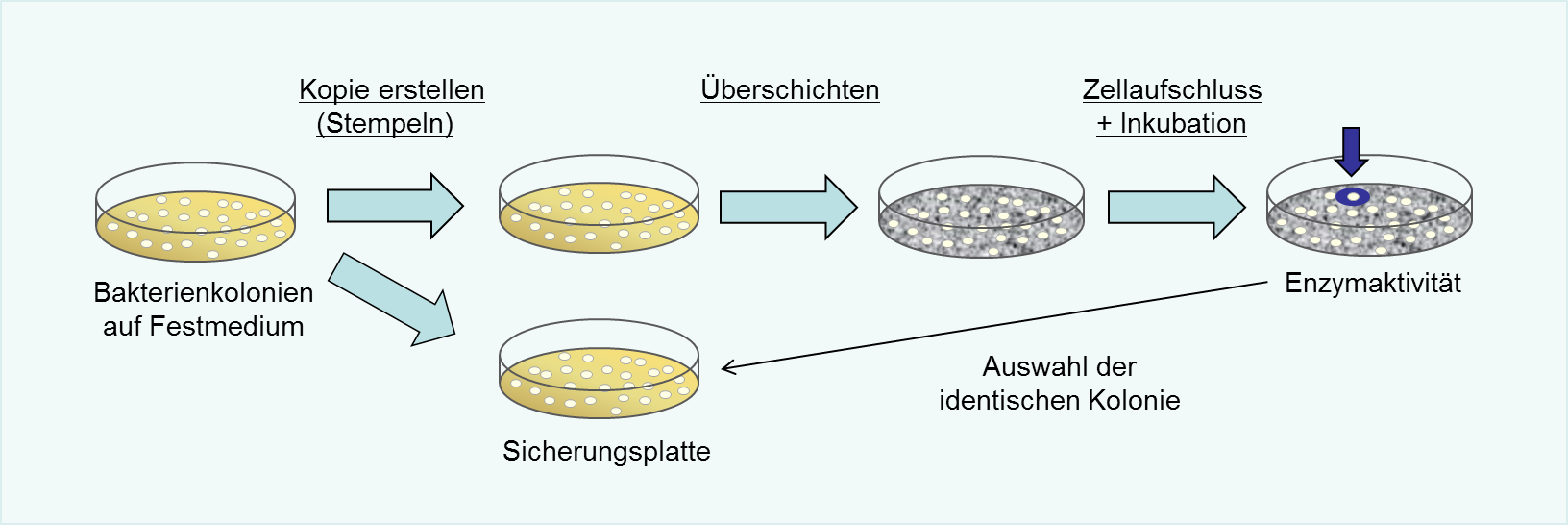
Jetzt wird es spannend: Wir müssen nun die Kolonie ausfindig machen, die unser gewünschtes Enzym produziert. Die Bakterien der Kolonie enthalten auf dem Plasmid den Bauplan für das Enzym - also das gesuchte Gen.
Für den Nachweis der Enzymaktivität muss das Enzym aus den Zellen freigesetzt werden. Dafür werden die Zellen aufgebrochen und zerstört. Das kann zum Beispiel durch Hitze erfolgen. Bei der Suche nach thermostabilen Enzymen bietet sich das an, da das Bakterium E. coli optimal bei 37 °C wächst, aber bei Hitze aufplatzt. Bei hohen Temperaturen „arbeitet“ auch das gesuchte thermostabile Enzym am besten. E. coli-Zellen können auch enzymatisch aufgebrochen werden. Dabei werden die Bakterienkolonien mit bestimmten Enzymen behandelt, wodurch die Zellwand und -membran der Bakterien zerstört und der Zellinhalt freigesetzt wird. E. coli-Zellen lysieren in ihrer finalen Phase des Wachstums (Absterbephase) nach einigen Tagen auf Kulturplatten auch von selbst (Autolyse). Dadurch kann unser gewünschtes Enzym auch freigesetzt werden.
Aber Achtung: Da wir am Ende lebendige Bakterien mit unserem gesuchten Gen benötigen, müssen wir vorher jeweils eine Kopie von unseren Kulturplatten erzeugen. Dies funktioniert wie beim Stempeln (siehe Video unten).
Abb.: Aktivitätsbasiertes Screening einer Genbank mit AZCL-Polysacchariden
Für den Nachweis von Enzymaktivitäten auf Kulturplatten gibt es verschiedene Methoden:
Zum Nachweis von Polysaccharid-abbauenden Enzymen, wie Cellulasen, werden zum Beispiel AZCL-Substrate verwendet (AZCL = azurine-cross-linked = Azurin-gekoppelt). Bei der AZCL-Cellulose ist das wasserunlösliche Polysaccharid mit dem blauen Farbstoff Azurin chemisch verbunden. AZCL-Polysaccharide bestehen daher aus kleinen wasserunlöslichen blauen Krümelchen. Auf der Suche nach Polysaccharid-abbauenden Enzymen wird eine Kulturplatte mit Bakterienkolonien mit dem entsprechenden AZCL-Polysaccharid überschichtet. An Stellen mit enzymatischer Aktivität wird das Polysaccharid in wasserlösliche Produkte gespalten, die in der Kulturplatte diffundieren können. Dadurch bildet sich eine blaue „Wolke“ um den Krümel. Diese Wolke nennen wir Halo.
Auch Fett-spaltende Enzyme, wie Lipasen und Esterasen, können in einer Metagenombank gesucht werden. Hierfür wird dem Nährmedium Tributyrin zugefügt. Da Tributyrin wasserunlöslich ist, wird die Kulturplatte milchig trüb. Nun werden die Bakterienzellen der Metagenombank darauf ausplattiert. Durch lipolytische Aktivität von Enzymen wird das Tributyrin in die wasserlöslichen Produkte Glycerin und Buttersäure gespalten. Die Kulturplatten werden an diesen Stellen klar und durchsichtig. Bakterienkolonien, die eine Lipase oder Esterase produzieren, weisen daher einen durchsichtigen Bereich um die Kolonie auf. Diesen Bereich nennen wir Halo.
Phytat ist das Salz der Phytinsäure und kommt in größeren Mengen in Getreide und Hülsenfrüchten vor. Es enthält sehr viele Phosphat-Ionen, welche durch Enzyme, die Phytasen genannt werden, abgespalten werden können. Um Phytasen in einer Metagenombank nachzuweisen, wird den Kulturplatten Calcium-Phytat zugefügt. Ca-Phytat ist ein wasserunlösliches Salz was die Kulturplatten milchig-weiß färbt. Phytasen können das Ca-Phytat in wasserlösliche Produkte spalten. Dadurch werden die milchig-weißen Kulturplatten an Stellen mit Phytase-Aktivität klar und durchsichtig. Bakterienkolonien, die eine Phytase produzieren, weisen daher einen durchsichtigen Bereich um die Kolonie auf. Diesen Bereich nennen wir Halo.



Haben wir eine Kolonie mit einem Halo gefunden, müssen wir anschließend nur noch auf der Originalplatte die identische Kolonie mit lebenden Zellen suchen. Diese enthält das Plasmid mit unserem gesuchten Gen.